古遺傳學家藉由研究古代 DNA,復原了數萬年前尼安德塔人的基因組,還得知丹尼索瓦人,這種與許多人祖先曾經混血過,其他性質卻一問三不知的神秘古人類。

第一批古埃及木乃伊的基因組,終於在今年問世惹!圖/取自 Nature 影片〈Cat domestication: From farms to sofas〉
不過尼安德塔人、丹尼索瓦人等等曾經共存的古人類們,後來全部滅絕了,只剩智人一種獨存。智人從非洲家鄉,前進世界各地的過程,仍有許多不明瞭的地方。這也是古代 DNA 大顯身手的領域,上千個已經發表的古代基因組,讓我們對人類遷徙、混血、發展的歷史,有了更清晰的認識,也回答不少之前不太清楚的疑惑。當然,解決舊問題之後,也開創了新的議題。
前進美洲
美洲遺傳史,是目前了解相對透徹的一塊。靠著分析大批古代樣本,以及現代族群的基因組,遺傳學家建構出移民美洲的大致歷史。
歐洲人抵達美洲以前,除了住在北極區的居民以外,各地的原住民族群,不論是古代的肯納威克人、Anzick-1、印加人,或現在的馬雅人、阿帕契人,遺傳上主要都能追溯到同一群人,他們在冰河時期的 2 萬多年前,與亞洲東北方的族群分家,然後跨越那時能夠通行的白令陸橋,抵達美洲,接著分散到美洲各地。[1]
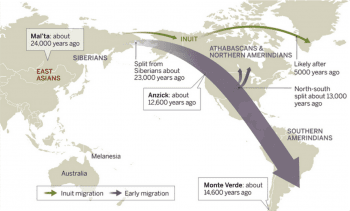
美洲移民的遺傳史。圖/取自〈New mystery for Native American origins〉
一個有趣的問題是,古時候的美洲移民何時南遷,又是怎麼南遷?南向的障礙是阻擋在北美洲西北方,冰河時期會融合為一體的勞倫斯冰蓋(Laurentide Ice Sheet)與柯迪勒拉冰蓋(Cordilleran Ice Sheet),要等到冰蓋退散,中間的通道露出以後才有機會走人。
為了回答這個問題,2 個研究團隊採取不同策略,得到類似卻略有不同的答案,顯示出用古代 DNA 解答問題的多樣性。
一樣的問題,用不同方法解答
一個研究由位於當年通道上的湖裡,取得沉積物,定序其中的「環境 DNA」。若是某年代的沉積物缺乏 DNA,意謂那時該地的環境無法維持動物生存,也不能讓人通過;等到沉積物中出現較多動物、植物的 DNA,人類才有通行的可能。判斷結果是,要等到距今 12600 年之後,通道才能夠走人。[2]
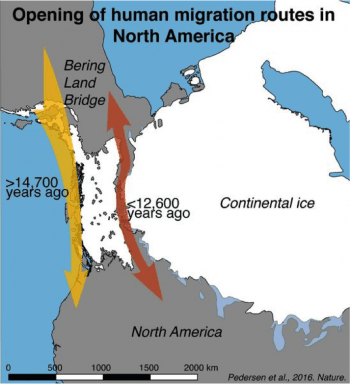
向美洲南部前進的障礙與通道。圖/取自〈Textbook story of how humans populated America is ‘biologically unviable,’ study finds〉
另一個研究則調查草原野牛(Bison priscus),南北兩地族群遺傳交流的狀況。野牛是大型動物,假如冰蓋封路,那麼南北族群間的遺傳交流會被中斷,等到冰蓋勢力退散,才會再度觀察到情慾流動;而野牛能走的路,人類大概也能通行。此一方法的估計是距今 13500 年前,比環境 DNA 判斷的年代更早一些。[3]
上述 2 個古代 DNA 研究,材料都不是人類,卻都能間接回答人類遷徙的問題。儘管兩種方法得到的年代有些差異,卻一致證實兩大冰蓋尚未退讓以前,人類已經先抵達美洲南方,否則智利不會存在超過 14000 年的遺址;而既然內陸行不通,能通過的路線,大概就是沿著海岸了。
農夫與他們的 DNA
冰河時期結束以後,中東肥沃月灣的人們發明農業,進入新石器時代,隨後新石器式的生活方式傳進歐洲。過往學者並不太肯定,到底農業是由移民引進歐洲,或是只涉及技術傳播?
至今最多古代基因組來自歐亞大陸西部,特別是歐洲,也讓上述問題有了明確的解答。比較中東及歐洲新石器時代的農夫,雙方族群的基因組相當類似,所以歐洲農業,來自由中東向歐洲遷徙的農夫。[4][5]
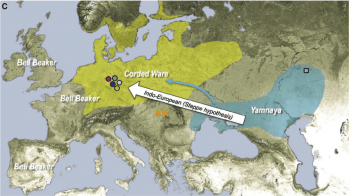
起源自東歐大草原的遊牧族群,將自己的 DNA 與印歐語,一起傳進歐洲。圖/取自〈Massive migration from the steppe was a source for Indo-European languages in Europe〉
在農夫移民以前,歐洲本來住著一些沒有農業,依靠採集狩獵維生的居民,他們後來上哪兒去了?比較歐洲各地,不同年代的古代基因組能看出,較早期的農夫基因組中,源自採集狩獵族群的比例很低,隨後數千年卻逐漸增加,意謂農夫抵達歐洲,在各地定居下來以後,漸漸與周遭的採集狩獵族群融合。[6]
距今 5000 年前,歐亞大陸西部開始轉型為青銅時代,馬、輪子的應用,拓展了人類長距離移動的能力,也造就史上最早的遊牧族群。那段時期的古代基因組顯示,源自東歐大草原的遊牧族群,DNA 同時影響東西兩邊,也就是中亞與歐洲 [7]。這波遷徙似乎也與印歐語系的傳播有關,不過同屬印歐語系的伊朗、印度,至今古代 DNA 樣本仍然有限,是有待未來探討的議題。
歐亞大陸西部的古代族群間,已知的一部份遺傳關係 @ @。圖/取自 ref 4
那些知名的古文化、古文明
古代 DNA 研究蓬勃發展,讓生命科學也加入探索歷史的行列。知名的古文明是由什麼人建立,能由古代 DNA 解答。例如歐洲青銅時代,發源於克里特島,全歐洲首度使用文字的米諾斯,以及隨後位於希臘的邁錫尼文明,當時居民的古代基因組,已經在今年發表。[8]
太平洋上的復活節島,以巨大的摩艾石像聞名;古時候島上居民的基因組,最近成功問世。要認識這個充滿神秘的古文化,古代 DNA 能提供難得的線索。[9]

復活節島上的摩艾石像。圖/取自〈 Paleogenomic analysis sheds light on Easter Island mysteries 〉
迦南人,是希伯來人的鄰居,住處現代稱作黎巴嫩。聖經描述他們遭到大難毀滅,但是由迦南人的古代基因組看來,聖經作者採用的應該是文創筆法,因為古迦南人的血脈,似乎仍有不少流傳到今日。[10]
不過眾多古文明中,大家最關心的大概還是古埃及。過往試圖取得古埃及 DNA 的嘗試,由於方法上的限制,結果都有些疑問。所幸隨著技術進步,第一批古埃及木乃伊的基因組,終於順利在今年發表。樣本年代介於新王國到羅馬時期,也就是所謂的「古埃及文明」後期;比較基因組發現,當時埃及人與同時期的中東鄰居-黎凡特人,遺傳上能夠視為同一種人。[11]
歷史記載中的病菌是?
古代 DNA 能回答的歷史之謎,還有疾病。西元 1519 年,中美洲的阿茲特克帝國被西班牙征服者擊敗,但是對中美洲居民更致命的打擊,是 1545 與 1576 年發生的 2 次「cocoliztli」,估計造成 700 到 1800 萬人死亡。歷史學家光靠文獻記載,難以確認如此兇殘的「病菌」究竟是誰。最近古代 DNA 研究,指出兇手應該是傷寒(不過該論文尚未正式發表)。[12][13]
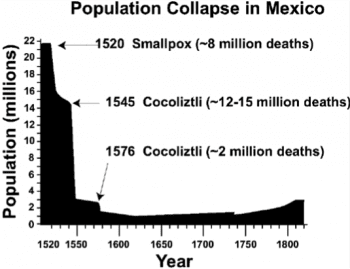
墨西哥歷年人口估計。圖/取自 wiki
近幾年由古代樣本定序得到 DNA 的微生物,還有結核菌、鼠疫桿菌、胃幽門螺旋桿菌、天花等等。其中研究最多的是鼠疫桿菌,它們以中世紀時造成的黑死病最為有名;而古代 DNA 研究則發現,早在 5000 年前,已經有人感染過這種疾病了。[14]
馴化、野生、滅絕的生物們
人類能夠適應世界各地,眾多的馴化生物功不可沒,藉由研究它們的古代 DNA,也能更加了解人類的歷史。發源於美洲的糧食作物玉米 [15],以及中東的大麥 [16],都已經有古代基因組發表;不過至今馴化動物的研究數目超過植物,較引人注目的有狗 [17] [18]、貓 [19]、馬 [20]、雞 [21]。

也有一些古代 DNA 研究,對象是野生動物,例如之前提過的草原野牛,還有真猛獁象(Mammuthus primigenius)、大西洋鱈等等。如真猛獁象、旅鴿(Ectopistes migratorius)般的滅絕生物,古代 DNA 能讓我們在化石型態以外,以另一種層面認識牠們失落的演化史。[22] [23] [24] [25]
也許沒那麼生物或演化,但是很文化
還有一些古代 DNA 研究,本身或許沒那麼有演化,或是生物的意義,卻比較偏向文化面。今年發表,丹麥古墓中菁英維京戰士的基因組,就是個好例子。DNA 分析清楚指出,擁有全套戰士裝備陪葬的墓主,生理上無疑是位女性;使得女生是否能稱為「戰士」,引發一陣討論熱潮。[26]
分析 DNA 當然只能回答墓主是不是女生,無法解決她是否為戰士的爭議;要解決這類問題,需要的是人類的智慧,而非遺傳物質。不過顯而易見,面對這種爭議性的敏感問題,古代 DNA 能提供比過往方法,更可靠的訊息。
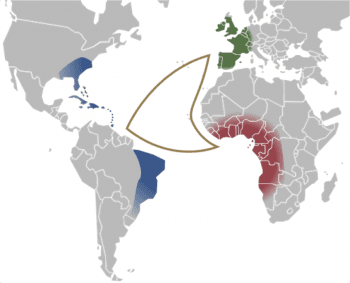
這個幾百年前,跨越大西洋的三角形貿易路線,稱作「中央通道(Middle Passage)」,其中一項主要商品,是非洲智人。圖/取自 wiki
人類歷史上有許多黑暗,古代 DNA 也許也能帶來一絲光明。在西元 1500 年到 1850 年間,史稱「大西洋奴隸貿易(Atlantic slave trade)」這段期間,超過 1200 萬非洲人被帶離家鄉,送往美洲各地為奴。這波規模極大的非自願人口遷徙,最終重新塑造了這個世界,以及定義「人類」的意義。
曾經有過這麼多人被迫離鄉背井,相遇、共處,然後一同埋骨異鄉。他們來自不同的文化背景,在新世界如何形塑新的文化,與身份認同?這是考古與文化人類學家不會錯過的問題,而古代遺傳學,對這些議題也能有貢獻。
- 延伸閱讀:短篇 古代DNA揭露的大西洋奴隸貿易史
曾經有 3 位 17 世紀時被解放的奴隸,沒能返回非洲家鄉,最後一同葬在加勒比海的聖馬丁島上。由基因組推測,三人應該分屬不同族群與文化背景,語言或許也不相通;那麼他們如何溝通,生前關係是什麼?和維京女戰士一樣,古代 DNA 本身無法解答問題,卻能提供更多證據。類似這樣的研究,仍持續進行中。[27] [28]
西元 1750 年繪製的聖海倫娜島。除了法蘭西大皇帝拿破侖以外,還有一些無名非洲人埋骨於此。圖/取自 ref 27
古代 DNA 給予我們更多機會
從帕波試圖取得木乃伊 DNA 到今天,已經過了 30 多年,世界和那時大大不同,研究古代 DNA 從異想天開的不務正業,變成尖端科學的熱門領域。最近幾年古代 DNA 的研究實在太多,本文篇幅有限,只能大致觸及幾點,卻仍有許多疏漏。
仍有太多有趣的問題等待回答,而古代 DNA 給了我們機會。比方說,台灣不少人對南島語族的歷史感到興趣,然而事實上不只南島語族,住在南亞、東南亞、東亞的漢藏、南亞、侗台等語族,至今仍沒有任何有關的古代基因組研究。這些人的歷史,也都很值得探討。還有一些問題,本來想都想像不到,例如我們的健康,竟然受到幾萬年前遠古混血的影響?
古代 DNA 在解答舊有疑惑的同時,也不斷開創新的問題,拓展人類智識的疆界。
延伸閱讀:
- 美洲原住民23000年來的族群歷史
- 艾斯克·威勒斯列夫,用基因重寫人類歷史
- 米諾斯人與邁錫尼人來自何方?走出古希臘神話,用DNA追追追
- 青銅時代3700年前迦南人,遺傳與現代黎巴嫩人高度一致
- 古代復活節島居民,沒有與美洲人交流DNA
- 木乃伊的 DNA (下):古今埃及人的變與不變
- 鼠疫桿菌:伴隨人類5000年的死神
- 新石器時代3個歐洲古狗基因組,仍有許多未解之謎
- 貓貓的世界征服史:從抓老鼠到沙發馬鈴薯
- 馬改變了人類文明,人又如何改變了馬?
- 雞排的起源?從古 DNA 探索雞的馴化與育種過程
- 維京的高階戰士不可能是女生?DNA分析能推翻十九世紀以來的偏見嗎?
- 航向太平洋的DNA之旅:南島語族與拉匹達關係解密
- 古代DNA帶我們認識東亞遺傳史
參考文獻:
- Raghavan, M., Steinrücken, M., Harris, K., Schiffels, S., Rasmussen, S., DeGiorgio, M., … & Eriksson, A. (2015). Genomic evidence for the Pleistocene and recent population history of Native Americans. Science, 349(6250), aab3884.
- Pedersen, M. W., Ruter, A., Schweger, C., Friebe, H., Staff, R. A., Kjeldsen, K. K., … & Potter, B. A. (2016). Postglacial viability and colonization in North America’s ice-free corridor. Nature, 537(7618), 45-49.
- Heintzman, P. D., Froese, D., Ives, J. W., Soares, A. E., Zazula, G. D., Letts, B., … & Jass, C. N. (2016). Bison phylogeography constrains dispersal and viability of the Ice Free Corridor in western Canada. Proceedings of the National Academy of Sciences, 201601077.
- Lazaridis, I., Nadel, D., Rollefson, G., Merrett, D. C., Rohland, N., Mallick, S., … & Connell, S. (2016). Genomic insights into the origin of farming in the ancient Near East. Nature, 536(7617), 419-424.
- Broushaki, F., Thomas, M. G., Link, V., López, S., van Dorp, L., Kirsanow, K., … & Kousathanas, A. (2016). Early Neolithic genomes from the eastern Fertile Crescent. Science, 353(6298), 499-503.
- Lipson, M., Szécsényi-Nagy, A., Mallick, S., Pósa, A., Stégmár, B., Keerl, V., … & Oppenheimer, J. (2017). Parallel palaeogenomic transects reveal complex genetic history of early European farmers. Nature.
- Mathieson, I., Lazaridis, I., Rohland, N., Mallick, S., Patterson, N., Roodenberg, S. A., … & Sirak, K. (2015). Genome-wide patterns of selection in 230 ancient Eurasians. Nature, 528(7583), 499-503.
- Lazaridis, I., Mittnik, A., Patterson, N., Mallick, S., Rohland, N., Pfrengle, S., … & McGeorge, P. J. P. (2017). Genetic origins of the Minoans and Mycenaeans. Nature, 548(7666), 214-218.
- Fehren-Schmitz, L., Jarman, C. L., Harkins, K. M., Kayser, M., Popp, B. N., & Skoglund, P. (2017). Genetic Ancestry of Rapanui before and after European Contact. Current Biology, 27(20), 3209-3215.
- Haber, M. et al. (2017) Continuity and admixture in the last five millennia of Levantine history from ancient Canaanite and present-day Lebanese genome sequences. Am. J. Hum. Genet.
- Schuenemann, V. J., Peltzer, A., Welte, B., van Pelt, W. P., Molak, M., Wang, C. C., … & Teßmann, B. (2017). Ancient Egyptian mummy genomes suggest an increase of Sub-Saharan African ancestry in post-Roman periods. Nature communications, 8, 15694.
- Collapse of Aztec society linked to catastrophic salmonella outbreak
- Vågene, Å. J., Campana, M. G., García, N. M. R., Warinner, C., Spyrou, M. A., Valtueña, A. A., … & Krause, J. (2017). Salmonella enterica genomes recovered from victims of a major 16th century epidemic in Mexico. bioRxiv, 106740.
- Rasmussen, S., Allentoft, M. E., Nielsen, K., Orlando, L., Sikora, M., Sjögren, K. G., … & Nielsen, H. B. (2015). Early divergent strains of Yersinia pestis in Eurasia 5,000 years ago. Cell, 163(3), 571-582.
- Swarts, K., Gutaker, R. M., Benz, B., Blake, M., Bukowski, R., Holland, J., … & Ross-Ibarra, J. (2017). Genomic estimation of complex traits reveals ancient maize adaptation to temperate North America. Science, 357(6350), 512-515.
- Mascher, M., Schuenemann, V. J., Davidovich, U., Marom, N., Himmelbach, A., Hübner, S., … & Schreiber, M. (2016). Genomic analysis of 6,000-year-old cultivated grain illuminates the domestication history of barley. Nature genetics.
- Frantz, L. A., Mullin, V. E., Pionnier-Capitan, M., Lebrasseur, O., Ollivier, M., Perri, A., … & Tresset, A. (2016). Genomic and archaeological evidence suggest a dual origin of domestic dogs. Science, 352(6290), 1228-1231.
- Botigué, L. R., Song, S., Scheu, A., Gopalan, S., Pendleton, A. L., Oetjens, M., … & Bobo, D. (2017). Ancient European dog genomes reveal continuity since the Early Neolithic. Nature Communications, 8, ncomms16082.
- Ottoni, C., Van Neer, W., De Cupere, B., Daligault, J., Guimaraes, S., Peters, J., … & Becker, C. (2017). The palaeogenetics of cat dispersal in the ancient world. Nature Ecology & Evolution, 1(7), 0139.
- Librado, P., Gamba, C., Gaunitz, C., Der Sarkissian, C., Pruvost, M., Albrechtsen, A., … & Serres-Armero, A. (2017). Ancient genomic changes associated with domestication of the horse. Science, 356(6336), 442-445.
- Loog, L., Thomas, M. G., Barnett, R., Allen, R., Sykes, N., Paxinos, P. D., … & Larson, G. (2017). Inferring Allele Frequency Trajectories from Ancient DNA Indicates That Selection on a Chicken Gene Coincided with Changes in Medieval Husbandry Practices. Molecular Biology and Evolution, msx142.
- Rogers, R. L., & Slatkin, M. (2017). Excess of genomic defects in a woolly mammoth on Wrangel island. PLoS genetics, 13(3), e1006601.
- Star, B., Boessenkool, S., Gondek, A. T., Nikulina, E. A., Hufthammer, A. K., Pampoulie, C., … & Petereit, C. (2017). Ancient DNA reveals the Arctic origin of Viking Age cod from Haithabu, Germany. Proceedings of the National Academy of Sciences, 201710186.
- Hung, C. M., Shaner, P. J. L., Zink, R. M., Liu, W. C., Chu, T. C., Huang, W. S., & Li, S. H. (2014). Drastic population fluctuations explain the rapid extinction of the passenger pigeon. Proceedings of the National Academy of Sciences, 111(29), 10636-10641.
- Murray, G. G., Soares, A. E., Novak, B. J., Schaefer, N. K., Cahill, J. A., Baker, A. J., … & Gilbert, M. T. P. (2017). Natural selection shaped the rise and fall of passenger pigeon genomic diversity. Science, 358(6365), 951-954.
- Hedenstierna‐Jonson, C., Kjellström, A., Zachrisson, T., Krzewińska, M., Sobrado, V., Price, N., … & Storå, J. (2017). A female Viking warrior confirmed by genomics. American Journal of Physical Anthropology.
- What DNA reveals about St Helena’s freed slaves
- Schroeder, H., Ávila-Arcos, M. C., Malaspinas, A. S., Poznik, G. D., Sandoval-Velasco, M., Carpenter, M. L., … & Samaniego, J. A. (2015). Genome-wide ancestry of 17th-century enslaved Africans from the Caribbean. Proceedings of the National Academy of Sciences, 112(12), 3669-3673.
本文亦刊載於作者部落格《盲眼的尼安德塔石匠》暨其 facebook 同名專頁。
The post 啊~ 追著你的人、追著你從哪來、追著你的發展歷史——古代DNA追追追(下) appeared first on PanSci 泛科學.